Coronavirus
Orthocoronavirinae · Orthocoronavirinés, Orthocoronavirus
| Realm | Riboviria |
|---|---|
| Règne | Orthornavirae |
| Embranchement | Pisuviricota |
| Classe | Pisoniviricetes |
| Ordre | Nidovirales |
| Sous-ordre | Cornidovirineae |
| Famille | Coronaviridae |
Genres de rang inférieur
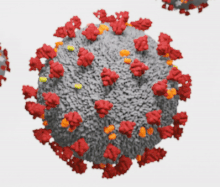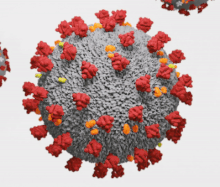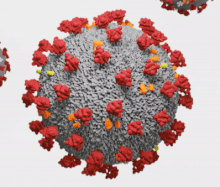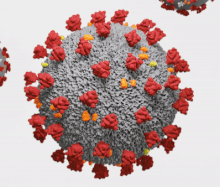
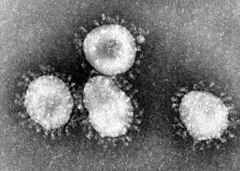
Les orthocoronavirus (CoV) sont des virus qui constituent la sous-famille Orthocoronavirinae de la famille Coronaviridae (les coronavirus). Le nom « coronavirus », du latin signifiant « virus à couronne », est dû à l'apparence des virions sous un microscope électronique, avec une frange de grandes projections bulbeuses qui évoquent une couronne solaire[2].
Ces coronavirus sont munis d'une enveloppe virale incluant une capside caractérisée par des protéines en forme de massue (appelées spicules). Ils ont un génome à ARN monocaténaire (c'est-à-dire à un seul brin), de sens positif (groupe IV de la classification Baltimore), de 26 à 32 kilobases (ce qui en fait les plus grands génomes parmi les virus à ARN)[3]. Ils se classent parmi les Nidovirales, ordre de virus produisant un jeu imbriqué d'ARNm sous-génomiques lors de l'infection. Des spicules, une enveloppe, membrane et capside contribuent à la structure d'ensemble de tous les coronavirus. Ils peuvent muter et se recombiner[4].
Les chauves-souris et les oiseaux, en tant que vertébrés volants à sang chaud, seraient les hôtes idéaux pour les coronavirus assurant l'évolution et la dissémination du virus[5]. Ces coronavirus sont normalement spécifiques à un taxon animal comme hôte, mammifères ou oiseaux selon leur espèce ; mais ils peuvent parfois changer d'hôte à la suite d'une mutation. Leur transmission interhumaine se produit principalement par contacts étroits via des aérosols respiratoires générées par les éternuements, la toux ou la phonation. Plus de 500 types de coronavirus ont été isolées chez la chauve-souris et il existerait plus de 5 000 types de coronavirus[6].
Sept principaux coronavirus sont généralement cités comme pouvant contaminer l'humain[7]. Un huitième a été identifié : le B814[8] (le premier coronavirus humain identifié), mais cette souche semble ne plus circuler.
Quatre coronavirus en circulation sont considérés comme sources d'infection bénignes : 229E, NL63, OC43 et HKU1. Ils seraient la cause de 15 à 30 % des rhumes courants.
Plus récemment ont été identifiés trois types de coronavirus responsables de graves pneumopathies :
- le SARS-CoV, agent pathogène du syndrome respiratoire aigu sévère (SRAS) en 2002-2004 ;
- le MERS-CoV, celui du syndrome respiratoire du Moyen-Orient à partir de 2012 ;
- le SARS-CoV-2, celui de la maladie à coronavirus 2019 (Covid-19) apparue en Chine en 2019 et responsable d'une sévère pandémie depuis 2019.
Découverte, histoire modifier
Les coronavirus existent probablement depuis au moins des centaines de millions d'années, mais du point de vue de l'épidémiologie et de l'histoire médicale et en tant que zoonose c'est au XXIe siècle qu'ils ont pris de l'importance : « cinq des sept coronavirus humains ont été isolés au cours de ce siècle. Et malheureusement, les trois derniers sont entrés dans notre vie avec les craintes liées à une épidémie, une pandémie ou à la mort »[9].
C'est en 1930 aux États-Unis que la première maladie due à un coronavirus est observée, chez des volailles. L'année suivante, un médecin décrit dans un article la maladie qui cause une détresse respiratoire chez la poule et une diminution de la ponte et de la qualité des œufs. En 1937, l'agent infectieux, le virus de la bronchite infectieuse aviaire (IBV pour Infectious Bronchitis Virus) est isolé.[réf. nécessaire]
En 1946, un autre coronavirus est identifié, le Coronavirus de la gastro-entérite transmissible porcine (TGEV). Indépendamment, en 1949 à New York et 1951 à Londres, deux équipes découvrent le virus de l'hépatite murine chez une souris paralysée[10].
En 1965, le premier coronavirus infectant l'être humain (la souche B814) est découvert. Et rapidement, d'autres suivent : 229E en 1966 et OC43 en 1967[11], qui sont la cause de rhumes plus ou moins graves selon les personnes. L'année suivante, ils sont observés au microscope électronique par June Almeida et David Tyrrell qui mettent en évidence leur structure en couronne[12]. La relation est faite entre tous ces virus, et le terme de « coronavirus » est pour la première fois utilisé dans la revue Nature en 1968[2],[10].
Avec la découverte en 2018 de coronavirus infectant des amphibiens et classés dans une sous-famille à part (letovirus), les coronavirus infectant des amniotes (comme ses mammifères et des oiseaux) sont classées dans la sous-famille Orthocoronavirinae[1] et qualifiés plus précisément d'orthocoronavirus[13].
Épidémie du XXIe siècle modifier
modifier
Le dernier coronavirus humain (ou récemment humanisé, très probablement à partir d'une ou plusieurs souches portées par des chauves-souris) semble avoir émergé à Wuhan en Chine en 2019 : le SARS-CoV-2. La maladie qu'il cause (Covid-19) a provoqué en quelques mois la première grande pandémie à coronavirus, caractérisée par un R0 élevé (2,3 en moyenne d'après les estimations disponibles en , mais qui semble pouvoir atteindre 5,7) ; avec un taux de létalité de 6,3 (très variable selon les âges et les contextes, pouvant parfois dépasser 15%)[9].
| Pandémie | Date | Cas confirmés | Décès | Guérisons | Sous-type impliqué |
|---|---|---|---|---|---|
| Épidémie de SRAS de 2002-2004 | 2002-2004 | 8 096 | 774 | - | Sars-Cov (SRAS) |
| Coronavirus du syndrome respiratoire du Moyen-Orient | 2012-2014 | 361 | 107 | - | MERS-CoV |
| Pandémie de Covid-19 (En cours) | 2019-2024 | + 250 847 494 ()[14] | + 5 064 350 ()[14] | + 220 905 435 ()[14] | SARS-CoV-2 (Covid-19) |
Caractérisation modifier
La taxonomie de ces nouveaux virus fait d'abord débat, pour finalement aboutir en 1975 à la création d'une nouvelle famille (Coronaviridae) et d'une nouvelle sous-famille (Orthocoronavirinae) par l'International Committee on Taxonomy of Viruses[10].
Dénomination modifier
Le terme coronavirus (du latin corona et virus, littéralement « virus à couronne »[15]) provient de l'apparence des virions au microscope électronique, caractérisée par une frange de grandes protubérances entourant l'enveloppe avec l'apparence d'une couronne, par analogie avec la couronne solaire[2].
Hôtes du virus modifier
Les hôtes idéaux des coronavirus, en tant que vertébrés volants à sang chaud, sont les chauves-souris (pour les Alphacoronavirus et les Betacoronavirus) et les oiseaux (pour les Gammacoronavirus et les Deltacoronavirus). Ces espèces-réservoir assurent l'évolution et la dissémination des coronavirus[5]. Chez d'autres espèces, les symptômes varient (maladies des voies respiratoires supérieures chez la poule, diarrhée chez la vache ou le porc, des voies digestives chez le chat et le chien, etc.). Parfois, aucun symptôme n'est associé à leur présence (ex. : coronavirus du béluga).
L'être humain abrite naturellement quatre types de coronavirus bénins, qui provoquent des infections des voies respiratoires, comme le rhume, et plus rarement affectent les systèmes gastro-intestinaux, cardiaques et nerveux[16].
Les groupes de coronavirus ont normalement un hôte animal spécifique (mammifères ou oiseaux[17]) mais ils peuvent parfois changer d'hôte à la suite d'une mutation. Ce sont de telles mutations qui ont probablement conduit à l'apparition de souches causant de graves infections chez l'homme (SRAS, MERS et Covid-19).
Tropisme modifier
On a longtemps pensé que les coronavirus avaient un tropisme uniquement respiratoire ou gastrointestinal (traduit par des pneumonies et entérocolites dans les cas graves), mais un nombre croissant d'études montrent un tropisme bien plus large, cardiovasculaire notamment, et neurologique également (dès les années 1980, on a montré que plusieurs coronavirus, dont en dernier cas le SARS-CoV-2 sont clairement aussi neuroinvasifs et neurotropes[18],[19],[20], au point que cette diversité de tropismes et de symptômes font des coronavirus (murins notamment, regroupées sous le sigle de MHV) un modèle animal pour l'étude de maladies humaines aussi variées que la sclérose en plaques, l’hépatite virale ou la pneumonie (S. R. Weiss et al. 2011). Le MHV pénètre le Système nerveux central (SNC) via les neurones du nerf olfactif, et peut causer une encéphalite aiguë ou une maladie démyélinisante chronique s'il y persiste (il peut aussi se propager jusqu’à la moelle épinière)[19],[21].
Recherches modifier
En 2002, l’apparition du Sars-CoV, un virus responsable d'une maladie infectieuse des poumons, pousse l’Union européenne à lancer plusieurs programmes afin de ne pas être prise au dépourvu en cas de nouvelles émergences. Dès 2004, l’équipe de Bruno Canard, directeur de recherche CNRS à Aix-Marseille, spécialiste des coronavirus, grâce aux réseaux collaboratifs européens, affiche des résultats prometteurs. « Nous avions eu cette idée qui s’est révélée fructueuse : les virus ont une capacité énorme à être différents, variés, avec de larges familles. Nous les avons donc étudiés tous en même temps, afin d’en avoir un modèle type qui nous permettrait, en cas de menace d’un virus inconnu, d’en trouver un proche, d’où nous pourrions extraire des données scientifiques[22]. »
Mais dès 2006, l’intérêt des politiques pour le Sars-CoV disparaît. La crise financière de 2008 accélère le désengagement de l’Europe et de la France pour la recherche, les stratégies de recherche fondamentale perdent leurs financements. Aussi, en 2015, Bruno Canard dénonce le désengagement européen et français dans le secteur des sciences et adresse avec ses collègues belges et hollandais, des lettres d’intention à la Commission européenne expliquant qu’il existe neuf familles de virus pour lesquelles une émergence est possible. « Le premier sur la liste était le flavivirus, explique-t-il. Le second, le coronavirus. Un an plus tard, apparaissait Zika, un flavivirus ». Or, la Commission européenne ne donnera jamais de réponse. Et en 2020 surgit le Sars-CoV-2, un coronavirus[22] engendrant la Covid-19.
Biologie modifier
Morphologie modifier
Ce virus enveloppé est constitué d'une enveloppe virale entourant une capside hélicoïdale qui contient le brin d'ARN. La taille du génome de ces virus varie d'environ 26 à 32 kilobases, valeurs parmi les plus élevées chez les virus à ARN.
Les coronavirus ont en commun des protéines désignées par une lettre indiquant leur localisation : S (protubérances), E (enveloppe), M (membrane) et N (nucléocapside). Certains, notamment ceux du sous-groupe A du genre Betacoronavirus, ont une protéine HE (hémagglutinine estérase (en)) caractéristique. Le coronavirus du SRAS présente en outre sur la protéine S un site de liaison spécifique à l'enzyme de conversion de l'angiotensine 2[23] qui lui sert de point d'entrée dans la cellule hôte.
La taille physique du virion est classiquement donnée comme étant de 120 à 160 nm[24] ou comme étant de l'ordre de 125 nm[25]. Toutefois le SARS-CoV-2, responsable de la Covid-19 a été annoncé plus récemment comme mesurant approximativement de 60 à 140 nm, et comme étant de forme elliptique avec de nombreuses variations[26].
Génome modifier
Tous les CoV ont un génome d'ARN non-segmentés (simple brin) organisé de la même manière : les deux tiers environ du génome contiennent deux grands « cadres de lecture ouverts » et se chevauchant (dits ORF1a et ORF1b). Ces deux cadres sont traduits en « polyprotéines réplicase » pp1a et pp1ab. « Ces polyprotéines sont ensuite traitées pour générer 16 protéines non structurales, désignées nsp1 ~ 16. La partie restante du génome contient des ORF pour les protéines structurales, y compris la pointe (S), l'enveloppe (E), la membrane (M) et la nucléoprotéine (N). Un certain nombre de protéines accessoires spécifiques à la lignée sont également codées par différentes lignées de CoV »[3],[27],[28],[29].
Réplication modifier
Elle se fait en six étapes successives (voir illustration) :
- Grâce à leur protéine S, les coronavirus se lient aux molécules cellulaires de surface telles que les métalloprotéinases. Les virus dotés en plus de la protéine HE (hémagglutinine-estérase) dans leur enveloppe peuvent aussi se lier à l'acide N-acétylneuraminique qui sert de corécepteur (lui-même initiateur de l'entrée d'un pathogène dans une cellule hôte). On ne sait pas clairement si les virus entrent dans la cellule hôte par fusion des membranes virales et cellulaires, ou par une internalisation à récepteur. Quel qu’en soit le mécanisme, le brin d'ARN est inséré dans la cellule, et la capside (la coque) est abandonnée ;
- Les coronavirus sont munis d'un seul génome ARN à brin positif, à présent sur place dans le cytoplasme. Le génome de l'ARN du coronavirus a une coiffe méthylée 5' et une queue polyadénylée 3', ce qui permet à l'ARN de se fixer aux ribosomes pour la traduction. Les ribosomes de la cellule décodent l'ARN viral, produisant les protéines qui y sont codées ;
- D'abord l'ARN positif du virus est transcrit en protéine pour former une ARN polymérase propre (une ARN polymérase ARN-dépendante). La réplicase est la première protéine fabriquée ; une fois le gène codant la réplicase traduit par le ribosome de la cellule hôte, la traduction est arrêtée par un codon stop. Cette réplicase virale ne reconnaît et produit que l'ARN viral, et permet au génome viral d'être transcrit en nouvelles copies d'ARN, à l'aide de la machinerie de la cellule hôte. Se servant du brin positif comme modèle, cet enzyme assemble le brin négatif ;
- Par la suite, ce brin négatif sert lui-même de modèle pour transcrire de petits ARN sous-génomiques, qui sont utilisés pour fabriquer toutes les autres protéines. C'est ce qu'on appelle une transcription imbriquée. Ce processus est une forme d'économie génétique, permettant au virus de coder le plus grand nombre de gènes dans un petit nombre de nucléotides ;
le génome du brin négatif est traduit par le ribosome de la cellule hôte, et une longue polyprotéine est formée, où toutes les protéines virales sont attachées. Les coronavirus ont une protéine non structurale — une protéase — qui est capable de cliver la polyprotéine.
Par ailleurs, ce brin négatif joue un rôle dans la réplication de nouveaux génomes ARN à brin positif.
Le cytoplasme de la cellule hôte se remplit de protéines et d'ARN viraux ; - (a) La protéine N aide à lier l'ARN génomique pour réaliser l’encapsidation du génome virale dans une enveloppe protectrice nommée capside[30] ; la protéine M s'intègre à la membrane du réticulum endoplasmique, côté capside ; et des protéines HE et S traversent la membrane du réticulum endoplasmique, via la protéine de translocation, et se positionnent du côté opposé ;
(b) avec la liaison entre la capside et les protéines M, la membrane du réticulum s'invagine, et bourgeonne. La capside (la coque) assemblée dotée d'ARN hélicoïdal se retrouve alors à l'intérieur du réticulum endoplasmique, ayant capturé à son profit la membrane de ce dernier, qui porte à présent à son extérieur les protéines HE et S ; - Cette progéniture virale est ensuite (a) encapsulée et transportée par des vésicules golgiennes vers la membrane cellulaire, (b) pour être enfin externalisée (par exocytose) hors de la cellule.
Taxonomie modifier
modifier
Les coronavirus sont nommés par un groupe d'étude[31] travaillant au sein de l'ICTV (International committee on Taxonomy of viruses)[32].
Classification modifier
Les coronavirus (CoV) sont des virus à ARN monocaténaire de sens positif (groupe IV de la classification Baltimore) correspondant à la sous-famille Orthocoronavirinae de la taxonomie de l'ICTV[33], dans la famille Coronaviridae, et de l'ordre Nidovirales[34],[35].
Selon les caractéristiques de leurs séquences protéiques, les CoV sont classés en 4 genres (alpha-CoV, beta-CoV, gamma-CoV et delta-CoV), qui tous contiennent des virus pathogènes pour les mammifères[4] :
- Alphacoronavirus, qui inclut le virus de la diarrhée épidémique porcine (PEDv), le virus de la gastro-entérite transmissible porcine (TGEV), le coronavirus du syndrome de la diarrhée aiguë porcine (SADS-CoV), le coronavirus canin, le coronavirus entérique félin, le virus de la péritonite infectieuse féline (FIPV) ;
- Betacoronavirus, dont le virus respiratoire du SRAS (SARS-CoV), le SARS-CoV-2, le coronavirus du syndrome respiratoire du Moyen-Orient (MERS-CoV), virus de l'hépatite murine (MHV), coronavirus bovins, virus de la sialodacryoadénite du rat, virus de la sialodacryoadénite porcine, hémagglutinose porcine, virus de l'hémagglutinose porcine coronavirus équin. Dans ce genre Betacoronavirus, le SARS-CoV et le SARS-CoV-2 appartiennent tous les deux au sous-genre Sarbecovirus au sein duquel trois clades distincts ont été identifiés :
- Clade1: souches "chauve-souris" de Bulgarie et Kenya[36],
- Clade2: SARS-CoV-2 et souches "chauve-souris" de Chine orientale[36],
- Clade3: SARS-CoV et souches "chauves-souris" de Chine du sud-ouest[36] ; - Gammacoronavirus: surtout trouvé chez des oiseaux migrateurs, causant notamment des bronchites ; un Gammacoronavirus a été isolé d'un béluga en captivité ;
- Deltacoronavirus: connus depuis peu, qui semblent surtout infecter les oiseaux, mais aussi trouvé chez les porcs.
Remarques :
- on a parfois nommé un coronavirus selon l'espèce animale où il a d'abord été trouvé (par exemple : coronavirus respiratoire canin, ou CRCoV pour Canine respiratory coronavirus, virus appartenant au genre betacoronavirus et à son sous-groupe 2a)[37],[38] ;
- le dernier coronavirus trouvé, en 2019, est le SARS-CoV-2, responsable de la pandémie de Covid-19.
Liste des espèces modifier
La sous-famille Orthocoronavirinae de la famille Coronaviridae est organisée en 4 genres, 22 sous-genres et une quarantaine d'espèces[39] :
- Sous-famille Orthocoronavirinae
- Genre Alphacoronavirus
- Sous-genre Colacovirus
- Bat coronavirus CDPHE15
- Sous-genre Decacovirus
- Bat coronavirus HKU10
- Rhinolophus ferrumequinum alphacoronavirus HuB-2013
- Sous-genre Duvinacovirus
- Human coronavirus 229E (HCoV-229E)
- Sous-genre Luchacovirus
- Lucheng Rn rat coronavirus
- Sous-genre Minacovirus
- Ferret coronavirus
- Mink coronavirus 1
- Sous-genre Minunacovirus
- Miniopterus bat coronavirus 1
- Miniopterus bat coronavirus HKU8
- Sous-genre Myotacovirus
- Myotis ricketti alphacoronavirus Sax-2011
- Nyctacovirus
- Nyctalus velutinus alphacoronavirus SC-2013
- Sous-genre Pedacovirus
- Porcine epidemic diarrhea virus
- Scotophilus bat coronavirus 512
- Sous-genre Rhinacovirus
- Rhinolophus bat coronavirus HKU2
- SADS-CoV (Coronavirus du syndrome de la diarrhée aiguë porcine)
- Rhinolophus bat coronavirus HKU2
- Sous-genre Setracovirus
- Human coronavirus NL63 (HCoV-NL63)
- NL63-related bat coronavirus strain BtKYNL63-9b
- Sous-genre Tegacovirus
- Alphacoronavirus 1
- Sous-genre Colacovirus
- Genre Betacoronavirus
- Sous-genre Embecovirus
- Betacoronavirus 1
- HCoV-OC43
- Bovine coronavirus
- Canine respiratory coronavirus
- Coronavirus HKU23
- Equine coronavirus
- Porcine hemagglutinating encephalomyelitis virus
- Yak coronavirus
- China Rattus coronavirus HKU24
- Human coronavirus HKU1 (HCoV-HKU1)
- Murine coronavirus
- Myodes coronavirus 2JL14
- Betacoronavirus 1
- Sous-genre Hibecovirus
- Bat Hp-betacoronavirus Zhejiang2013
- Sous-genre Merbecovirus
- Hedgehog coronavirus 1
- Middle East respiratory syndrome-related coronavirus (MERS-CoV)
- Pipistrellus bat coronavirus HKU5
- Tylonycteris bat coronavirus HKU4
- Sous-genre Nobecovirus
- Rousettus bat coronavirus GCCDC1
- Rousettus bat coronavirus HKU9
- Sous-genre Sarbecovirus
- Severe acute respiratory syndrome-related coronavirus (SARSr-CoV)
- SARS-CoV (humain; SRAS)
- SARSr-CoV WIV1 (chauve-souris)
- SARSr-CoV HKU3 (chauve-souris)
- SARSr-CoV RP3 (chauve-souris)
- SARS-CoV-2 (humain; Covid-19)
- Severe acute respiratory syndrome-related coronavirus (SARSr-CoV)
- Sous-genre Embecovirus
- Genre Gammacoronavirus
- Sous-genre Cegacovirus
- Beluga whale coronavirus SW1
- Sous-genre Igacovirus
- Sous-genre Cegacovirus
- Genre Deltacoronavirus
- Sous-genre Andecovirus
- Wigeon coronavirus HKU20
- Sous-genre Buldecovirus
- Bulbul coronavirus HKU11
- Coronavirus HKU15
- Munia coronavirus HKU13
- White-eye coronavirus HKU16
- Sous-genre Herdecovirus
- Night heron coronavirus HKU19
- Sous-genre Moordecovirus
- Common moorhen coronavirus HKU21
- Sous-genre Andecovirus
- Genre Alphacoronavirus
modifier
Types d'infection modifier
Sept types de coronavirus infectent couramment l'homme[40], dont trois causent des infections graves.
Infections bénignes modifier
Les quatre premiers types connus sont sans gravité : les coronavirus humains 229E, NL63, OC43 et HKU1, inconnus chez la chauve-souris. Ils causent des rhumes avec fièvre et des maux de gorge dus à des végétations adénoïdes gonflées, principalement en hiver et au début du printemps[41].
Les coronavirus seraient la cause de 15 à 30 % des rhumes courants[42].
Infections graves modifier
Trois types de coronavirus qui ne se trouvent pas naturellement chez l'homme mais chez des mammifères ont été découverts plus récemment et ont été à l'origine d'infections graves des poumons (pneumopathie virale) :
- le SARS-CoV, agent pathogène du syndrome respiratoire aigu sévère (SRAS) dont l'épidémie de 2002-2004 a déclenché une alerte mondiale de l'OMS. Elle a débuté en Chine après la consommation dans un restaurant d'un animal sauvage, la civette palmiste masquée. La maladie a fait 774 morts (10 % environ des personnes atteintes). Elle est considérée comme éradiquée depuis 2004 ;
- le MERS-CoV, celui du syndrome respiratoire du Moyen-Orient dont la première épidémie a débuté en Arabie saoudite en 2012. Son taux de mortalité a été de 35 %, faisant [Quand ?]449 victimes seulement du fait du faible nombre d'individus atteints. [réf. nécessaire]Elle aurait été déclenchée par la consommation de lait de chameau et par la proximité avec les chameaux. Cette maladie existe toujours car pour pouvoir l'éradiquer, il faudrait que les populations qui utilisent traditionnellement des chameaux puissent s'en passer ;
- le SARS-CoV-2, celui de la maladie à coronavirus 2019 (Covid-19) apparue en Chine en 2019 et responsable d'une sévère pandémie en 2020. La consommation de viande de pangolin[43] et de chauve-souris (vendues pour l'alimentation en Chine) ; ou un virus crée dans un laboratoire en Chine [44],[45] pourraient en être à l'origine.
Selon le virus en cause, les formes graves de la maladie ont leurs particularités. Par exemple, la diarrhée était très fréquente dans le SRAS mais rare dans la maladie à coronavirus 2019.
Comparaison des infections graves modifier
Trois principales sources sont utilisées : l'Institut Pasteur, l'OMS et les CDC américains[46].
| Syndrome respiratoire du Moyen-Orient (MERS)[47],[48] | Syndrome respiratoire aigu sévère (SRAS)[49],[50],[51] | Maladie à coronavirus 2019 (COVID-19) | |
|---|---|---|---|
| Agent pathogène | MERS-CoV | SARS-CoV | SARS-CoV-2 |
| Année d'apparition | 2012 | 2003 | 2019 |
| Nombre de cas | 1 219 | 8 098 dont 5 327 en Chine. | En cours, voir ici |
| Pourcentage de cas par transmission nosocomiale | 70 %[52] | 58 %[52] | |
| Nombre de décès | 449 | 774 (dont 349 en Chine) | En cours, voir ici |
| Réservoir | Dromadaire | Chauve-souris | Chauve-souris (probablement) |
| Transmission à l'homme par l'animal | Contact direct avec un animal infecté, consommation de lait cru de dromadaire. | Consommation de viande de civette palmiste masquée, animal sauvage vendu sur les marchés et consommé dans le Sud de la Chine. | Un pangolin[43] pourrait être l'hôte intermédiaire. |
| Transmission interhumaine | Oui | Oui | Oui |
| Transmission par objet | – | Oui | Risque très faible. |
| Transmission materno-fœtale | – | Aucun cas retrouvé chez les femmes enceintes infectées par ce virus[53]. | Aucune preuve[54]. |
| Transmission par le lait maternel | Un seul cas documenté[55] | RT-PCR négative sur 16 femmes testées[56] | |
| Incubation | Entre 5 et 15 jours. | Entre 2 et 7 jours. | Durée médiane d'incubation à 5,1 jours (5,5 jours en moyenne), 97,5 % des personnes seront malades 11,5 jours après le contact infectieux[57]. |
| Porteur sain | – | Probablement pas. | Oui (un seul cas publié) |
| Contagiosité | Taux de reproduction inférieur à 1[58]. | Taux de reproduction supérieur à 2[58]. | Médiane du taux de reproduction de base (R0) à 2,79[59]. |
| Durée de la contagiosité | – | – | Semble limitée à la période des signes cliniques. Possibilité disputée de contagion en phase asymptomatique[60]. |
| Début de la période de contagiosité | – | 3 à 4 jours après le début des signes cliniques. | Dès l'apparition des signes cliniques. Porteur asymptomatique prouvé[61]. |
| Fièvre | À 98% | À 99% | À 87.9%, mais peut apparaître plusieurs jours après la toux ou les difficultés à respirer. |
| Diarrhée | À 26% | À 20%[53]. | À 3.7%. |
| Transmission par les selles | Très probable mais a joué un rôle mineur[53]. | Cette possibilité est envisagée[62]. | |
| Létalité | 34,4 %[52] | 9,5 %[52], au-delà de 50 % chez les plus de 65 ans. | 3,4 %[63] |
| Traitement | Symptomatique | Symptomatique | Symptomatique |
| Vaccin | Aucun | Aucun | En cours |
| Statut | Résurgence possible. | Considéré comme éradiqué. | Épidémie en cours. |
Passage de la barrière des espèces modifier
Au vu des séquences génomiques disponibles, deux grands taxons animaux seraient le réservoir principal des CoVs :
- chiroptères : hôtes naturels du HCoV-NL63 et du HCoV-229E[64] ;
- rongeurs : hôtes naturels du HCoV-OC43 et HKU1[64].
Au vu des connaissances disponibles, les coronavirus semblaient avoir besoin d'hôtes intermédiaires (toujours des mammifères) pour s'« humaniser », c'est-à-dire muter pour pouvoir infecter l'Homme.
Des hôtes intermédiaires connus ont été :
- des bovins pour HCoV-OC43[64] ;
- l'alpaga pour HCoV-229E[64] ;
- la civette masquée pour le SARS-CoV[64] ;
- le dromadaire pour le MERS-CoV[64].
Transmission interhumaine modifier
La transmission interhumaine des coronavirus se fait principalement par les gouttelettes ou des aérosols respiratoires expectorées par une personne infectée (via la toux, les éternuements, des postillons, ou parfois par le simple fait de parler fort ou en criant) quand les particules virales sont inhalées par une personne se trouve à proximité. La transmission et la contagiosité varient aussi selon le coronavirus, et peut-être selon sa souche au sein d'une épidémie.
La prophylaxie passe par une prévention primaire visant à limiter la transmission du virus : éviter les contacts (surfaces potentiellement contaminées, poignées de main, embrassades), se laver les mains fréquemment, éviter de se toucher les yeux, le nez ou la bouche, par où le virus peut s'introduire dans l'organisme. En cas de symptômes de type toux ou rhume, se maintenir à au moins 1 mètre de toute personne et éviter d'émettre des particules contaminées[65].
D'autres recommandations comprennent[66] :
- ne pas entrer en contact avec des animaux manifestement malades, ne pas consommer de viandes provenant d'animaux malades ;
- ne pas consommer de produits animaux (viande...) mal cuits, ni de légumes crus s'ils n'ont pas été lavés avec de l'eau non contaminée.
Traitement modifier
Dans le cas du SRAS, des médicaments ont été utilisés pour tenter d'enrayer l'épidémie : la ribavirine, un analogue de nucléotides, des anti-inflammatoires stéroïdiens et, après identification formelle de l'agent pathogène et des criblages de sensibilité, l'interféron-alpha et des inhibiteurs de protéases. Leur efficacité est encore sujette à caution. Aucun n'a fait l'objet d'une étude clinique adéquate : beaucoup d'études disponibles ne permettent pas de conclusions scientifiques claires car elles ont été réalisées sur de petits nombres de sujets ou alors sans protocole ou dose fixe. Certaines indiquent même que ces traitements pourraient avoir nui à l'éradication du virus[67].
Bruno Canard dénonce en l'emballement et publie une lettre ouverte Coronavirus : la science ne marche pas dans l’urgence ![68]. Il déclare : « Un vaccin demande au mieux 18 mois de recherches. Et pour des virus non prévisibles, qui changent, il n’est pas adapté. Mieux vaut faire des médicaments qui ont un large spectre dans une famille virale. Cela peut nécessiter 5 ans, parfois 10. D’où l’importance de l’anticipation scientifique[22]. »
Vaccins modifier
Des vaccins à base de virus inactivé et d'autres fondés sur les protéines S et N sont à l'étude depuis plusieurs années[69] quand l'éradication rapide de l'épidémie de SRAS vient interrompre les essais cliniques. Cependant, les éléments viraux produisant l'immunité ne sont souvent pas assez conservés dans la même famille virale : « s'il y avait eu un vaccin contre le coronavirus de 2003, il est pratiquement certain qu'il n'aurait pas marché de manière satisfaisante contre [la] Covid-19 » (Bruno Canard)[70].
Bien que n'ayant pas abouti à leur certification, la recherche de vaccins contre le SRAS a rendu possible l'obtention rapide de vaccins contre la Covid-19. Ainsi, le vaccin de Pfizer et BioNTech est conçu en quelques heures dès [71], et autorisé au Royaume-Uni en décembre. En , 60 vaccins contre le SARS-CoV-2 sont autorisés ou en phase d'étude clinique, et 172 vaccins potentiels sont à l'étude[72].
En 2023, certains variants du SARS-CoV-2, et notamment le variant Omicron, échappent en partie aux différents vaccins administrés. Plusieurs équipes dans le monde cherchent à produire des vaccins conférant une immunité plus durable et plus efficace contre les variants existants et à venir, ainsi que contre d'autres coronavirus. Elles utilisent de nouvelles approches[73] : élargissement des cibles protéiniques et recherche de nouvelles cibles, emploi de nanoparticules, etc.
Dans la culture populaire modifier
Un Romain dans la bande dessinée Astérix se nomme Coronavirus.
Notes et références modifier
- ICTV. International Committee on Taxonomy of Viruses. Taxonomy history. Published on the Internet https://talk.ictvonline.org/., consulté le 1era oût 2023
- (en) « Virology: Coronaviruses », Nature, vol. 220, no 5168, , p. 650–650 (ISSN 0028-0836 et 1476-4687, PMCID PMC7086490, DOI 10.1038/220650b0, lire en ligne, consulté le )
- (en) Zi-Wei Ye et Shuofeng Yuan, « Zoonotic origins of human coronaviruses », sur International Journal of Biological Sciences, (ISSN 1449-2288, PMID 32226286, PMCID PMC7098031, DOI 10.7150/ijbs.45472, consulté le ), p. 1686–1697.
- SARS-CoV, B.S (2020) Mutation and Recombination.
- (en) Patrick C. Y. Woo, Susanna K. P. Lau, Carol S. F. Lam, Candy C. Y. Lau, Alan K. L. Tsang, John H. N. Lau, Ru Bai, Jade L. L. Teng, Chris C. C. Tsang, Ming Wang, Bo-Jian Zheng, Kwok-Hung Chan et Kwok-Yung Yuen, « Discovery of Seven Novel Mammalian and Avian Coronaviruses in the Genus Deltacoronavirus Supports Bat Coronaviruses as the Gene Source of Alphacoronavirus and Betacoronavirus and Avian Coronaviruses as the Gene Source of Gammacoronavirus and Deltacoronavirus », Journal of Virology, vol. 86, no 7, , p. 3995-4008 (PMID 22278237, DOI 10.1128/JVI.06540-11, Bibcode 3302495, lire en ligne, consulté le )
- « Coronaviruses 101: Focus on Molecular Virology » (consulté le ).
- (en-US) « Coronavirus | Human Coronavirus Types | CDC », sur cdc.gov, (consulté le ).
- A. Z. Kapikian, « The coronaviruses », Developments in Biological Standardization, vol. 28, , p. 42–64 (ISSN 0301-5149, PMID 1092577, lire en ligne, consulté le )
- Cemal BULUT et Yasuyuki KATO, « Epidemiology of COVID-19 », TURKISH JOURNAL OF MEDICAL SCIENCES, vol. 50, no SI-1, , p. 563–570 (ISSN 1303-6165, DOI 10.3906/sag-2004-172, lire en ligne, consulté le )
- K McIntosh, W B Becker et R M Chanock, « Growth in suckling-mouse brain of "IBV-like" viruses from patients with upper respiratory tract disease. », Proceedings of the National Academy of Sciences of the United States of America, vol. 58, no 6, , p. 2268–2273 (ISSN 0027-8424, PMID 4298953, lire en ligne, consulté le )
- Steven Brocklehurst, « The woman who discovered the first coronavirus », sur bbc.com, British Broadcasting Corporation, (consulté le ).
- (en) « Family: Coronaviridae », sur International Committee on Taxonomy of Viruses, (consulté le ).
- (en) « COVID-19 Dashboard by the Center for Systems Science and Engineering (CSSE) at Johns Hopkins University (JHU) », sur gisanddata.maps.arcgis.com (consulté le ).
- « S'informer sur le Coronavirus », sur popsciences.universite-lyon (consulté le ).
- Zhu N., Zhang D., Wang W. et al., « A Novel Coronavirus from Patients with Pneumonia in China, 2019. », The New England Journal of Medicine, vol. 382, no 8, , p. 727-733 (PMID 31978945, PMCID PMC7092803, DOI 10.1056/NEJMoa2001017).
- Jacobson E.R (2007) Infectious Diseases and Pathology of Reptiles; CRC Press, Taylor & Francis Group: Boca Raton, FL, États-Unis ; p. 33487–2742 (lien Google Livre)
- (en) K. Yagami, K. Hirai et N. Hirano, « Pathogenesis of haemagglutinating encephalomyelitis virus (HEV) in mice experimentally infected by different routes », Journal of Comparative Pathology, vol. 96, no 6, , p. 645–657 (PMID 3546411, PMCID PMC7130377, DOI 10.1016/0021-9975(86)90061-7, lire en ligne, consulté le )
- (en) Susan J. Bender et Susan R. Weiss, « Pathogenesis of Murine Coronavirus in the Central Nervous System », Journal of Neuroimmune Pharmacology, vol. 5, no 3, , p. 336–354 (ISSN 1557-1890 et 1557-1904, PMID 20369302, PMCID PMC2914825, DOI 10.1007/s11481-010-9202-2, lire en ligne, consulté le )
- (en) Timothy J Cowley et Susan R Weiss, « Murine coronavirus neuropathogenesis: determinants of virulence », Journal of NeuroVirology, vol. 16, no 6, , p. 427–434 (ISSN 1355-0284 et 1538-2443, DOI 10.1007/BF03210848, lire en ligne, consulté le )
- Lane, Thomas E et Martin P Hosking. 2010. « The pathogenesis of murine coronavirus infection of the central nervous system. » Critical reviews in immunology 30 (2): 119‐30.
- « Bruno Canard, le chercheur qui avait alerté en 2015 sur le risque de Coronavirus, dénonce le désengagement dans la recherche », sur L'Humanité, (consulté le ).
- (en) Fang Li, Wenhui Li, Michael Farzan et Stephen C. Harrison, « Structure of SARS Coronavirus Spike Receptor-Binding Domain Complexed with Receptor », Science, vol. 309, no 5742, , p. 1864-1868 (PMID 16166518, DOI 10.1126/science.1116480, JSTOR 3843779, Bibcode 2005Sci...309.1864L, lire en ligne, consulté le )
- (en) Thomas C. Holland, Neurovirology in Handbook of Clinical Neurology, 2014 lire en ligne
- (en) Fehr, Anthony R, Stanley Perlman. “Coronaviruses: an overview of their replication and pathogenesis.” Methods in molecular biology (Clifton, N.J.) vol. 1282 (2015): 1-23. doi:10.1007/978-1-4939-2438-7_1 lire en ligne
- Cascella M, Rajnik M, Cuomo A, et al. Features, Evaluation and Treatment Coronavirus (COVID-19), in: StatPearls [Internet], StatPearls Publishing, Treasure Island (Floride), janvier 2020, mis à jour le 8 mars 2020 lire en ligne
- (en) Shuo Su et Gary Wong, « Epidemiology, Genetic Recombination, and Pathogenesis of Coronaviruses », sur Trends in Microbiology, (DOI 10.1016/j.tim.2016.03.003, consulté le ), p. 490–502.
- (en) Lok-Yin Roy Wong et Pak-Yin Lui, « A molecular arms race between host innate antiviral response and emerging human coronaviruses », sur Virologica Sinica, (ISSN 1674-0769, PMID 26786772, PMCID PMC7090626, DOI 10.1007/s12250-015-3683-3, consulté le ), p. 12–23.
- (en) Shuo Su et Gary Wong, « Epidemiology, Genetic Recombination, and Pathogenesis of Coronaviruses », sur Trends in Microbiology, (PMID 27012512, PMCID PMC7125511, DOI 10.1016/j.tim.2016.03.003, consulté le ), p. 490–502.
- (en) Ping-Kun Hsieh, Shin C. Chang, Chu-Chun Huang et Ting-Ting Lee, « Assembly of severe acute respiratory syndrome coronavirus RNA packaging signal into virus-like particles is nucleocapsid dependent », Journal of Virology, vol. 79, no 22, , p. 13848–13855 (ISSN 0022-538X, PMID 16254320, PMCID 1280188, DOI 10.1128/JVI.79.22.13848-13855.2005, lire en ligne, consulté le ).
- coronavirus Study Group ou CSG
- Gorbalenya A.E & a. (2020) Severe acute respiratory syndrome-related coronavirus: The species and its viruses – a statement of the Coronavirus Study Group. bioRxiv, https://www.biorxiv. org/content/10.1101/2020.02.07.937862v1.full.pdf.
- (en) « Virus Taxonomy: 2018b Release », ICTV, (consulté le ).
- de Groot RJ, Baker SC, Baric R, Enjuanes L, Gorbalenya AE, Holmes KV, Perlman S, Poon L, Rottier PJ, Talbot PJ, Woo PC, Ziebuhr J, Ninth Report of the International Committee on Taxonomy of Viruses, Oxford, Elsevier, , 806–828 p. (ISBN 978-0-12-384684-6), « Family Coronaviridae ».
- International Committee on Taxonomy of Viruses, « ICTV Master Species List 2009 – v10 » [xls], .
- Lu R, Zhao X, Li J, Niu P, Yang B, Wu H, [...] Tan W, 2020. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. The Lancet doi: https://doi.org/10.1016/S0140-6736(20)30251-8.
- (en) Kerstin Erles, Kai-Biu Shiu et Joe Brownlie, « Isolation and sequence analysis of canine respiratory coronavirus », Virus Research, vol. 124, no 1, , p. 78–87 (ISSN 0168-1702, DOI 10.1016/j.virusres.2006.10.004, lire en ligne, consulté le ).
- (en) Kerstin Erles, Crista Toomey, Harriet W Brooks et Joe Brownlie, « Detection of a group 2 coronavirus in dogs with canine infectious respiratory disease », Virology, vol. 310, no 2, , p. 216–223 (ISSN 0042-6822, DOI 10.1016/S0042-6822(03)00160-0, lire en ligne, consulté le ).
- (en) « Virus taxonomy: 2018b release », sur ICTV online, (consulté le ).
- (en) CDC, « Human Coronavirus Types », sur Center for Disease Control Prevention (consulté le ).
- (en) Liu P, Shi L, Zhang W, He J, Liu C, Zhao C, Kong SK, Loo JF, Gu D, Hu L, « Prevalence and genetic diversity analysis of human coronaviruses among cross-border children », Virology Journal, vol. 14, no 1, , p. 230 (PMID 29166910, PMCID 5700739, DOI 10.1186/s12985-017-0896-0).
- Fehr AR, Perlman S, « Coronaviruses: an overview of their replication and pathogenesis », Methods in Molecular Biology, vol. 1282, , p. 1–23 (ISBN 978-1-4939-2437-0, PMID 25720466, PMCID 4369385, DOI 10.1007/978-1-4939-2438-7_1).
- Julie Kern, « Le SARS-CoV-2 serait un mélange de coronavirus de pangolin et de chauve-souris », sur Futura (consulté le ).
- « Nouveaux doutes sur l’origine du Covid-19 - C à vous - 07/06/2022 » (consulté le ).
- « Covid-19. La thèse d’une fuite d’un laboratoire chinois mérite d’être approfondie, selon l’OMS ».
- Coronavirus Disease (COVID-19 cdc.gov, consulté le 02 mars 2020.
- « MERS-CoV », Institut Pasteur, (consulté le ).
- (en-US) CDC, « Middle East Respiratory Syndrome (MERS) », sur Centers for Disease Control and Prevention, (consulté le ).
- « SRAS », Institut Pasteur, (consulté le ).
- O.M.S, « Syndrome aiguë respiratoire »(Archive.org • Wikiwix • Archive.is • Google • Que faire ?), sur O.M.S (consulté le ).
- (en-US) « SARS | Home | Severe Acute Respiratory Syndrome | SARS-CoV Disease | CDC », sur cdc.gov, (consulté le ).
- Vincent J. Munster, Marion Koopmans, Neeltje van Doremalen et Debby van Riel, « A Novel Coronavirus Emerging in China — Key Questions for Impact Assessment », The New England Journal of Medicine, vol. 382, no 8, , p. 692–694 (ISSN 0028-4793, DOI 10.1056/NEJMp2000929, lire en ligne, consulté le ).
- Organisation mondiale de la santé, "Consensus document on the epidemiology of severe acute respiratory syndrome (SARS)", 2003, page 15.
- (en) Huijun Chen, Juanjuan Guo, Chen Wang et Fan Luo, « Clinical characteristics and intrauterine vertical transmission potential of COVID-19 infection in nine pregnant women: a retrospective review of medical records », The Lancet, vol. 395, no 10226, , p. 809–815 (ISSN 0140-6736 et 1474-547X, PMID 32151335, DOI 10.1016/S0140-6736(20)30360-3, lire en ligne, consulté le ).
- Corwin A. Robertson, Sara A. Lowther, Thomas Birch et Christina Tan, « SARS and Pregnancy: A Case Report », Emerging Infectious Diseases, vol. 10, no 2, , p. 345–348 (ISSN 1080-6040 et 1080-6059, PMID 15030710, PMCID PMC3322896, DOI 10.3201/eid1002.030736, lire en ligne, consulté le )
- (en) Kimberly A. Lackey, Ryan M. Pace, Janet E. Williams et Lars Bode, « SARS-CoV-2 and human milk: what is the evidence? », medRxiv, , p. 2020.04.07.20056812 (DOI 10.1101/2020.04.07.20056812, lire en ligne, consulté le )
- Étude du Johns Hopkins Bloomberg School of Public Health, de Baltimore associée à School of Public Health and Health Sciences du Massachusetts, et à la Ludwig-Maximilians-Universität de Munich.
- (en) David L. Swerdlow et Lyn Finelli, « Preparation for Possible Sustained Transmission of 2019 Novel Coronavirus: Lessons From Previous Epidemics », JAMA, (ISSN 0098-7484, DOI 10.1001/jama.2020.1960, lire en ligne, consulté le ).
- « Le taux de reproduction de COVID-19 est plus élevé que celui du SRAS ».
- « Coronavirus : une affiche du ministère écarte trop vite le risque de contagion lors de l’incubation », sur Le Monde, (consulté le ).
- (en) Smriti Mallapaty, « What the cruise-ship outbreaks reveal about COVID-19 », Nature, , d41586–020–00885-w (ISSN 0028-0836 et 1476-4687, DOI 10.1038/d41586-020-00885-w, lire en ligne, consulté le ).
- « Coronavirus : la maladie pourrait aussi se transmettre par voie fécale », sur pourquoidocteur.fr (consulté le ).
- « Coronavirus : L’OMS indique que le taux de mortalité est de 3,4 % », Intellivoire, (lire en ligne, consulté le ).
- (en) Jie Cui, Fang Li et Zheng-Li Shi, « Origin and evolution of pathogenic coronaviruses », Nature Reviews Microbiology, vol. 17, no 3, , p. 181–192 (ISSN 1740-1534, DOI 10.1038/s41579-018-0118-9, lire en ligne, consulté le ).
- Coronavirus disease (COVID-19) advice for the public, OMS, 2020.
- « Coronavirus – informations et conseils », sur Centre de vaccinations internationales Air France (consulté le ).
- Stockman LJ, Bellamy R, Garner P. SARS: systematic review of treatment effects. PLoS Med. 2006;3:e343. doi:10.1371/journal.pmed.0030343
- « Coronavirus : la science ne marche pas dans l’urgence ! », (consulté le ).
- Zhu, M. 2004. SARS immunity and vaccination. Cell. Mol. Immunol. 1:193-198
- Filippi 24 mars 2021 à 17 h 32 min Répondre, « Bruno Canard : «Demander un médicament dès le lendemain d’une épidémie n’a aucun sens» – Sciences Critiques » (consulté le ).
- (en) Susie Neilson, « The co-founder of BioNTech designed the coronavirus vaccine it made with Pfizer in just a few hours over a single day », sur Business Insider France, (consulté le ).
- (en) « Draft landscape of COVID-19 candidate vaccines ».
- Ewen Callaway, « La prochaine génération de vaccins contre les coronavirus », Pour la science, no 546, , p. 66-72.
Voir aussi modifier
Bibliographie modifier
- (en) Christine V.F. Carrington, Jerome E. Foster, Hua Chen Zhu, Jin Xia Zhang, Gavin J.D. Smith, Nadin Thompson, Albert J. Auguste, Vernie Ramkissoon, Abiodun A. Adesiyun et Yi Guan, « Detection and Phylogenetic Analysis of Group 1 Coronaviruses in South American Bats », Emerging Infectious Diseases journal, Centers for Disease Control and Prevention, vol. 14, no 12, , p. 1890-1893 (DOI 10.3201/eid1412.080642, lire en ligne)
- Habibzadeh P, Stoneman EK "The Novel Coronavirus: A Bird's Eye View". The International Journal of Occupational and Environmental Medicine. 11 (2): 65–71. doi:10.15171/ijoem.2020.1921. (résumé).
- Saif, L. J., Wang, Q., Vlasova, A. N., Jung, K., & Xiao, S. (2019). Coronaviruses. Diseases of swine, 488-523.
- Nathalie Kin et Astrid Vabret, « Les infections à coronavirus humains », sur Revue Francophone des Laboratoires, (PMID 32288826, PMCID PMC7140280, DOI 10.1016/S1773-035X(16)30369-0, consulté le ), p. 25–33
Infographie modifier
- Frédérique Schneider, « Coronavirus, les chiffres de l’épidémie », sur La Croix, (consulté le )
Articles connexes modifier
- Coronavirus du syndrome respiratoire du Moyen-Orient (MERS-CoV pour Middle East respiratory syndrome coronavirus, anciennement NCoV)
- Virologie
- Coronavirus félin
- Péritonite infectieuse féline
- Maladie à coronavirus 2019
- Syndrome respiratoire aigu sévère
- Pneumonie aiguë
- Urgence de santé publique de portée internationale (USPPI)
- Sociétés biopharmaceutiques en développement sur un vaccin : Moderna Therapeutics, CureVac, etc.
- Pandémie de Covid-19
Liens externes modifier
- (en) Référence ICTV : Orthocoronavirinae (consulté le )
- Ressources relatives à la santé :
- Notices dans des dictionnaires ou encyclopédies généralistes :
- (en) CDC, CDC Novel Coronavirus et CDC’s case definitions and guidance.
- (en) CDC, Site internet (CDC) consacré au novel coronavirus.
- (en) OMS, WHO Novel Coronavirus Infection—UpdateExternal.
- (en) Health Protection Agency Novel Coronavirus 2012.
- (fr) Cartes situation générale Cartograf.fr : Carte du Coronavirus COVID-19.
- (en) ECDC, ECDC: Novel Coronavirus update to Rapid Risk AssessmentExternal.
- (en) CDC Mise à jour : Severe Respiratory Illness Associated with a Novel Coronavirus—Worldwide, 2012–2013 MMWR March 7, 2013/62 (Early Release), p. 1-2.
- (en) CDC Severe Respiratory Illness Associated with a Novel Coronavirus — Saudi Arabia and Qatar, 2012 MMWR October 12, 2012/61, p. 820-820.
- Thankyoucaretakers.com/fr, initiative mise en place lors de la pandémie de COVID-19 de 2019/2020 et ayant pour objectif de rassembler 1 million de messages de remerciements pour les soignants du monde.