Groupes interne et externe (cladistique)
En cladistique ou en phylogénétique, un groupe interne (ou endogroupe ; en anglais : ingroup) correspond à l'ensemble des organismes à l'étude dont les caractères sont considérés proches, de telle sorte à former par opposition un groupe externe dont les caractères sont considérés comme plus éloignés.
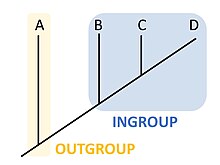
En cladistique ou en phylogénétique, un groupe externe (ou extra-groupe ; en anglais : outgroup)[1] est un ensemble d'organismes qui sert de groupe de référence pour déterminer les relations évolutives d'un groupe interne. Son rôle est de servir de point de comparaison pour le groupe interne et permet ainsi de créer un arbre phylogénétique enraciné. Parce que la polarité (direction) du changement des caractères ne peut être déterminée que sur une phylogénie enracinée, le choix du groupe externe est essentiel pour comprendre le sens évolutif des organismes[2].
Histoire modifier
Bien que le concept de groupe externe ait été utilisé depuis les premiers jours de la cladistique, le terme « outgroup » aurait été inventé au début des années 1970 au Musée américain d'histoire naturelle[3]. Avant l'avènement du terme, divers autres adjectifs étaient utilisés par les biologistes évolutionnistes, notamment « exgroupe », groupe apparenté (en anglais : related group) et groupe extérieur (en anglais : outside groups)[3].
Choix du groupe externe modifier
Le groupe externe choisi est supposé être moins étroitement lié au groupe interne que le groupe interne n'est lié à lui-même. La conclusion évolutionniste de ces relations est que l'espèce du groupe externe a un ancêtre commun avec le groupe interne qui est plus ancien que l'ancêtre commun du groupe interne. Le choix du groupe externe peut fortement affecter la topologie d'une phylogénie[4]. Par conséquent, les phylogénéticiens utilisent généralement plus d'un groupe externe dans l'analyse cladistique.
En effet, l'utilisation de plusieurs groupes externes est souvent préférable. Cela permet de fournir une phylogénie plus robuste, en médian les effets des moins bons candidats du groupe externe, et permet de tester la monophylie hypothétique du groupe interne[3],[5],[6].
Pour être qualifié de groupe externe, un taxon doit satisfaire les deux caractéristiques suivantes :
- Il ne doit pas être membre du groupe interne.
- Il doit être lié au groupe interne de manière assez étroite pour pouvoir réaliser des comparaisons significatives avec ce dernier.
Par conséquent, un groupe externe approprié doit être en dehors du clade d'intérêt de l'étude phylogénétique. Utiliser un groupe externe qui est trop proche du groupe interne pour enraciner un arbre phylogénétique conduit à des conclusions incorrectes sur les relations phylogénétiques et l'évolution des traits[7]. De plus, le niveau optimal de parenté du groupe externe et du groupe interne dépend de la profondeur de l'analyse phylogénétique. Choisir un groupe externe étroitement lié au groupe interne est utile lorsque l'on examine des différences subtiles, tandis qu'un groupe externe trop éloigné peut lui conduire à confondre une évolution convergente par une relation évolutive directe due à un ancêtre commun[8],[9].
Pour une analyse phylogénétique superficielle (comme la résolution des relations évolutives d'un clade au sein d'un genre), le groupe externe approprié est un membre du clade frère[10]. Cependant, pour une analyse phylogénétique plus approfondie, des taxons moins étroitement apparentés peuvent être utilisés. Par exemple, Jarvis et al. (2014) ont utilisé des humains et des crocodiles comme groupes externes pour résoudre les premières branches de la phylogénie aviaire[11]. En phylogénétique moléculaire, un bon groupe externe doit avoir ses séquences ADN ou protéiques alignées sur les séquences du groupe interne. Bien qu'il existe des approches algorithmiques pour identifier les groupes externes (méthode de parcimonie globale maximale), elles sont souvent limitées en ne reflètent pas la nature continue et quantitative de certains états des caractères[12]. En effet, les états des caractères sont des traits, ancestraux ou dérivés, qui affectent la construction de motifs de ramification dans un arbre phylogénétique[13].
Exemples modifier
| Groupe interne | Groupe externe |
|---|---|
| Grands singes [14] | Gibbons |
| Mammifères placentaires [15] | Marsupiaux |
| Chordés [16] | Échinodermes |
| Angiospermes [17] | Gymnospermes |
Dans chaque exemple, la phylogénie du groupe interne peut être enracinée en notant les caractères semblables pour un ou plusieurs membres du groupe externe.
Voir également modifier
- Apomorphie, un trait dérivé d'un organisme
- Groupe frère, un groupe qui peut être étroitement lié à un endogroupe
- La plésiomorphie, trait ancestral d'un organisme
Notes et références modifier
- David Grimaldi, Michael S. Engel et Michael S. Engel, Evolution of the Insects, (ISBN 9780521821490, lire en ligne)
- Farris, « Outgroups and Parsimony », Systematic Biology, vol. 31, no 3, , p. 328–334 (ISSN 1063-5157, DOI 10.1093/sysbio/31.3.328)
- Nixon et Carpenter, « On Outgroups », Cladistics, vol. 9, no 4, , p. 413–426 (DOI 10.1111/j.1096-0031.1993.tb00234.x)
- Giribet et Ribera, « The position of arthropods in the animal kingdom: a search for a reliable outgroup for internal arthropod phylogeny », Molecular Phylogenetics and Evolution, vol. 9, no 3, , p. 481–488 (PMID 9667996, DOI 10.1006/mpev.1998.0494)
- Barriel et Tassy, « Rooting with Multiple Outgroups: Consensus Versus Parsimony », Cladistics, vol. 14, no 2, , p. 193–200 (DOI 10.1111/j.1096-0031.1998.tb00332.x)
- de la Torre-Barcena, Kolokotronis, Lee et Stevenson, « The Impact of Outgroup Choice and Missing Data on Major Seed Plant Phylogenetics Using Genome-Wide EST Data », PLOS ONE, vol. 4, no 6, , e5764 (PMID 19503618, PMCID 2685480, DOI 10.1371/journal.pone.0005764, Bibcode 2009PLoSO...4.5764D)
- Maddison et al., « Outgroup Analysis and Parsimony », Systematic Zoology, vol. 33, no 1, , p. 83–103 (DOI 10.2307/2413134, JSTOR 2413134, lire en ligne)
- Wilberg, « What's in an Outgroup? The Impact of Outgroup Choice on the Phylogenetic Position of Thalattosuchia (Crocodylomorpha) and the Origin of Crocodyliformes », Systematic Biology, vol. 64, no 4, , p. 621–637 (ISSN 1063-5157, PMID 25840332, DOI 10.1093/sysbio/syv020)
- O'BRIEN, LYMAN, SAAB et SAAB, « Two Issues in Archaeological Phylogenetics: Taxon Construction and Outgroup Selection », Journal of Theoretical Biology, vol. 215, no 2, , p. 133–150 (PMID 12051970, DOI 10.1006/jtbi.2002.2548)
- David A. Baum et Stacey D. Smith, Tree Thinking: An Introduction to Phylogenetic Biology, Roberts, (ISBN 978-1-936221-16-5, lire en ligne), p. 175
- Jarvis et al., « Whole-genome analyses resolve early branches in the tree of life of modern birds », Science, vol. 346, no 6215, , p. 1320–1331 (PMID 25504713, PMCID 4405904, DOI 10.1126/science.1253451, Bibcode 2014Sci...346.1320J)
- Stevens, « Character States, Morphological Variation, and Phylogenetic Analysis: A Review », Systematic Botany, vol. 16, no 3, , p. 553–583 (DOI 10.2307/2419343, JSTOR 2419343)
- Rineau, Grand, Zaragüeta et Laurin, « Experimental systematics: sensitivity of cladistic methods to polarization and character ordering schemes », Contributions to Zoology, vol. 84, no 2, , p. 129–148 (DOI 10.1163/18759866-08402003)
- Prado-Martinez et Marques-Bonet, « Great ape genetic diversity and population history », Nature, vol. 499, no 7459, , p. 471–475 (PMID 23823723, PMCID 3822165, DOI 10.1038/nature12228, Bibcode 2013Natur.499..471P)
- Murphy, Pringle, Crider et Springer, « Using genomic data to unravel the root of the placental mammal phylogeny », Genome Research, vol. 17, no 4, , p. 413–421 (PMID 17322288, PMCID 1832088, DOI 10.1101/gr.5918807)
- Cameron, Garey et Swalla, « Evolution of the chordate body plan: New insights from phylogenetic analyses of deuterostome phyla », PNAS, vol. 97, no 9, , p. 4469–4474 (PMID 10781046, PMCID 18258, DOI 10.1073/pnas.97.9.4469)
- Matthews et Donoghue, « The Root of Angiosperm Phylogeny Inferred from Duplicate Phytochrome Genes », Science, vol. 286, no 5441, , p. 947–950 (PMID 10542147, DOI 10.1126/science.286.5441.947)