Gélose Chapman
La gélose Chapman ou MSA (de l'anglais Mannitol Salt Agar) est un milieu de culture sélectif et différentiel employé en microbiologie pour la culture des bactéries halophiles et halotolérantes. Il a été mis au point en 1945 par G.H. Chapman[1]. Grâce à son indicateur coloré qui détecte la consommation du mannitol, il est adapté à la recherche des Staphylocoques d'intérêt médical et notamment du pathogène Staphylococcus aureus (Staphylococque doré) dans toutes sortes de prélèvements cliniques et environnementaux.
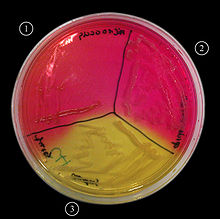

Principe modifier
La base nutritive se compose de peptone, d'extrait de bœuf et de D-mannitol.
La sélectivité du milieu provient de sa forte teneur en chlorure de sodium : s'élevant à 7,5% soit 75 g/L, elle lui confère un caractère hypertonique marqué. Seuls les organismes halophiles ou franchement halotolérants peuvent s'y développer ce qui élimine un grand nombre de contaminants (la plupart des Entérocoques, des Streptocoques et des bactéries Gram négatives dont les Entérobactéries[2]).
Le rouge de phénol est un indicateur de pH de couleur rouge orenagée dans la gamme de pH initiale du milieu (7,4 ± 0,2). Il permet la lecture du caractère mannitol : les bactéries capable de métaboliser ce monosaccharide le dégradent en sous-produits acides qui s'accumulent dans le milieu et entraînent un virage au jaune de l'indicateur. Les autres bactéries ne consomment que la peptone, produisant des déchets basiques qui font virer l'indicateur au rouge.
Lecture modifier
Les colonies suspectes de Staphylocoque doré sont celles qui présentent le caractère mannitol (colonies « mannitol + ») c'est-à-dire qu'elles jaunissent le milieu environnant.
Même si ce caractère peut avoir une valeur d'orientation il ne permet que de suspecter la présence de S. aureus. La croissance d'une colonie faisant virer au jaune le milieu Chapman montre seulement qu'il s'agit d'un micro-organisme halotolérant à 7,5% de chlorure de sodium capable de dégrader le mannitol en formant des sous-produits acides. D'autres tests sont nécessaires pour confirmer – ou non – l'identification[3],[4].
Composition modifier
Pour 1000 mL de milieu[5] :
- peptone : 10 g
- extrait de bœuf : 1 g
- chlorure de sodium : 75 g
- D-mannitol : 10 g
- rouge de phénol : 25 mg
- agar : 15 g.
Ajuster le pH à 7,4 ± 0,2 à 25°C (après autoclavage).
Selon les fabricants, les 10 g/L de peptone quelconque peuvent être remplacés par une combinaison de 5 g/L de peptone pancréatique de caséine et 5 g/L de peptone pepsique de viande.
Préparation modifier
Aucun ingrédient n'étant thermosensible, il suffit de les dissoudre à chaud dans le volume correspondant d'eau distillée et de stériliser à l'autoclave (15 minutes à 121°C). Le mélange est ensuite réparti dans des contenants stériles.
Notes et références modifier
- Chapman GH « The Significance of Sodium Chloride in Studies of Staphylococci » J Microbiol. 1945 Aug; 50(2):201-3. Accès libre.
- https://www.bd.com/resource.aspx?IDX=8827, consulté le 31/12/20.
- En particulier la coloration de Gram, la recherche d'une catalase et d'une coagulase, voire d'une thermonucléase etc.
- Kateete DP et al. « Identification of Staphylococcus aureus: DNase and Mannitol salt agar improve the efficiency of the tube coagulase test » Ann Clin Microbial Antimicrob. 2010 Aug; 9:23. Accès libre.
- http://www.himedialabs.com/TD/M118.pdf, consulté le 31/12/20.