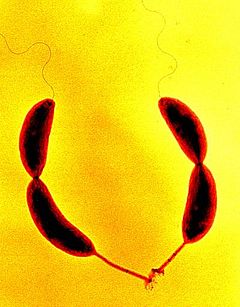
Caulobacter vibrioides
Caulobacter crescentus
| Domaine | Bacteria |
|---|---|
| Sous-règne | Negibacteria |
| Embranchement | Proteobacteria |
| Classe | Alphaproteobacteria |
| Ordre | Caulobacterales |
| Famille | Caulobacteraceae |
| Genre | Caulobacter |
Caulobacter vibrioides (anc. Caulobacter crescentus)[5] est une espèce de bactéries gram-négatives vivant dans les milieux aquatiques comme les lacs et les rivières. C’est une bactérie oligotrophe capable de croître dans les habitats pollués, grâce à des gènes impliqués dans la résistance aux métaux de transition et aux stress oxydants[6],[7].
Cette bactérie se présente sous deux formes très différentes : une forme non réplicative mobile grâce à un flagelle et une forme réplicative fixée munie d'un pédoncule. Alors que la forme mobile permet la dissémination et la recherche de nutriments, la forme fixée est le siège de la réplication. Lorsque les conditions sont favorables, la forme mobile se différencie vers la forme fixée pour se diviser de manière asymétrique et polarisée : une cellule mère fixée se divise alors en une cellule fille fixée et une cellule fille mobile[7].
C. vibrioides est utilisée comme modèle dans l’étude de la régulation du cycle cellulaire procaryote, de la division asymétrique et de la différenciation cellulaire. Le pédoncule confère à la cellule fixée une propriété particulière, l'adhésion, qui met en jeu des macromolécules appelées adhésines à structure polysaccharidique[8].
Deux souches modifier
Il existe deux souches de Caulobacter crescentus : la souche originelle CB15 et la souche utilisée en laboratoire NA1000. NA1000 est une souche dérivée de CB15 associée à une pression de sélection induit par l’environnement du laboratoire. Au niveau de la taille du génome, la souche CB15 fait 4,02 mégabases et la souche NA1000 fait 4,04 mégabases. Les deux souches sont génétiquement différentes par la présence de 8 polymorphismes codants, 2 non codants et 1 site d’insertions/suppressions (26 kb)[9].
Ces différences génétiques ont induit des différences phénotypiques entre les deux souches notamment au niveau de l’adhérence, la vitesse de croissance et la synchronisation. La synchronisation est la capacité de séparer physiquement les deux formes cellulaires, flagellées et pédonculées par centrifugation. Alors que la souche CB15 est adhérente (forme fixée), se multiplie lentement et est incapable de se synchroniser, la souche NA1000 n’est pas adhérente, se multiplie plus rapidement et possède la capacité de synchronisation. Cette dernière caractéristique fait de la souche NA1000, la souche prédominante en laboratoire[9].
Cycle cellulaire modifier
La forme flagellée mobile, permettant la dispersion, est limitée à la phase G1 de l'interphase. En conditions favorables, la bactérie se différencie en forme pédonculée fixée et permet la réplication du chromosome en phase S. Après la réplication du chromosome, la cellule s'allonge et s'apprête à se diviser en phase G2. La division cellulaire est asymétrique et permet de générer une cellule fixée qui va de nouveau répliquer le chromosome, et une cellule mobile qui va permettre la dissémination dans l'environnement. L'initiation de la réplication, la ségrégation des chromosomes et la cytokinèse constituent les trois grandes étapes du cycle cellulaire[7].
Étapes du cycle cellulaire modifier
La première étape, l'initiation de la réplication ne se fait que lorsque la cellule est en phase S quand l'origine de réplication, ori, est accessible. En plus d'être un régulateur transcriptionnel, la protéine CtrA régule le cycle cellulaire : lorsque celle-ci est phosphorylée, elle se lie au niveau de la région ori et inhibe la réplication du chromosome. La protéine CtrA phosphorylée est fortement présente dans les cellules flagellées, puis est déphosphorylée et dégradée après la différenciation en cellule pédonculée, permettant l'initiation de la réplication par la protéine DnaA[10].
Après la réplication, le chromosome originel et sa copie doivent être distribués aux deux cellules filles. Cette étape est appelée ségrégation des chromosomes et est régulée par le système de partition ParABS. parS est une séquence d'ADN située près de l'ori correspondant à un centromère où se fixe la protéine de fixation ParB. La protéine ParA se fixe au complexe et permet l'hydrolyse de l'ATP entraînant la migration des chromosomes d'une part et d'autre de la cellule bactérienne[11].
La cytokinèse, étape de division en deux cellules filles, débute par l'assemblage de l'anneau Z au centre de la cellule. L'anneau Z est constitué de la protéine FtsZ qui se polymérise et permet de générer les forces constriction essentielles à la division de la cellule. Afin que l'assemblage de l'anneau Z soit bien localisé, il est nécessaire que la polymérisation de FtsZ soit inhibée dans le reste de la cellule. Cette inhibition est exercée par la protéine MipZ recrutée par la protéine ParB créant un gradient de concentration : MipZ est plus important au niveau des pôles et moins important au centre de la cellule[12]. La séparation des deux cellules filles est assurées par plusieurs protéines : FtsEX, un transporteur de type ABC permet l'ancrage de la protéine FtsZ dans la membrane, FtsK, une translocase à ADN permet de transloquer l’ADN qui serait au niveau du septum et le complexe FtsQL permet la stabilité du divisome. Enfin, les protéines FtsN, FtsW et FtsI permettent la biosynthèse et le remodelage du peptidoglycane induisant ainsi la septation et la séparation des deux cellules filles[13].
Asymétrie du cycle cellulaire modifier
Le cycle cellulaire chez Caulobacter crescentus permet de générer deux formes cellulaires différentes. Au-delà de la différence structurale, l'assemblage excentré de l'anneau Z permet d'obtenir une cellule fille pédonculée légèrement plus grande que la cellule fille flagellée[7]. Cette asymétrie du cycle est intrinsèquement liée à l'accumulation de différentes protéines à chaque pôle de la cellule : on parle de polarité cellulaire[14].
Au niveau protéique, une des différences entre la cellule mobile et la cellule fixée est la concentration en CtrA phosphorylée, protéine régulant la réplication cellulaire. La phosphorylation de la protéine CtrA est contrôlée par un complexe de phosphorelais, dont une unité diverge entre la future cellule flagellée et la future cellule pédonculée. Basiquement, la phosphorylation de CtrA est initiée par la protéine régulatrice Ccka et passe par l'intermédiaire d'une protéine ChpT. L'activation de Ccka dépend de son interaction avec la protéine DivL, complexe qui peut être inhibée par la protéine DivK. Enfin, la phosphorylation de DivK est contrôlée par la phosphatase PleC au pôle de la future cellule mobile et par la kinase DivJ au pôle de la future cellule fixée. La protéine PleC désactive DivK, DivK n'inhibe plus Ccka-DivL, Ccka en tant que kinase est activée et permet la phosphorylation de CtrA, ce qui empêche l'initiation de la réplication dans la future cellule mobile. La protéine DivJ active DivK, DivK peut inhiber Ccka-DivL, Ccka permet la déphosphorylation de CtrA, ce qui permet l'initiation de la réplication dans la future cellule fixée[15].
La combinaison des protéines ZitP, PopZ et CpaM permet la distinction majeure entre les deux cellules : la présence d'un flagelle ou d'un pédoncule. La protéine doigt de zinc ZitP possède deux fonctions, dépendant du pôle de la cellule[14]. Au pôle de la future cellule mobile, ZitP recrute la protéine effectrice CpaM et permet l'assemblage du flagelle alors qu'au pôle de la future cellule fixée, ZitP s'associe à la protéine PopZ afin de contrôler sa position lors du cycle cellulaire. PopZ est une protéine présente dans les deux pôles de la cellule en division qui interagit directement avec le système ParABS[16]. L'association des protéines ZitP et CpaM au pôle de la future cellule mobile et non au pôle fixée est influencée par le système régulant la réplication cellulaire, DivJ-PleC-DivK[17].
Notes et références modifier
- (en) « Species: Caulobacter vibrioides », sur lpsn.dsmz.de (consulté le )
- Catalogue of Life Checklist, consulté le 14 juin 2021
- Brands, S.J. (ed.), 1989-present. The Taxonomicon. Universal Taxonomic Services, Zwaag, The Netherlands. [http://taxonomicon.taxonomy.nl/], consulté le 14 juin 2021
- Integrated Taxonomic Information System (ITIS), www.itis.gov, CC0 https://doi.org/10.5066/F7KH0KBK, consulté le 14 juin 2021
- (en) « Caulobacter vibrioides (strain NA1000 / CB15N) (Caulobacter crescentus) », sur www.uniprot.org (consulté le )
- (en) Christian Kohler, Rogério F Lourenço, Gabriela M Avelar et Suely L Gomes, « Extracytoplasmic function (ECF) sigma factor σF is involved in Caulobacter crescentus response to heavy metal stress », BMC Microbiology, vol. 12, no 1, , p. 210 (ISSN 1471-2180, PMID 22985357, PMCID PMC3511200, DOI 10.1186/1471-2180-12-210, lire en ligne, consulté le )
- « Impact du cuivre sur le cycle cellulaire de Caulobacter Crescentus », sur Portail de la recherche de l'Université de Namur (consulté le )
- (en) P. H. Tsang, G. Li, Y. V. Brun et L. B. Freund, « Adhesion of single bacterial cells in the micronewton range », Proceedings of the National Academy of Sciences, vol. 103, no 15, , p. 5764–5768 (ISSN 0027-8424 et 1091-6490, PMID 16585522, PMCID PMC1458647, DOI 10.1073/pnas.0601705103, lire en ligne, consulté le )
- (en) Melissa E. Marks, Cyd Marie Castro-Rojas, Clotilde Teiling et Lei Du, « The Genetic Basis of Laboratory Adaptation in Caulobacter crescentus », Journal of Bacteriology, vol. 192, no 14, , p. 3678–3688 (ISSN 0021-9193 et 1098-5530, PMID 20472802, PMCID PMC2897358, DOI 10.1128/JB.00255-10, lire en ligne, consulté le )
- Encyclopædia Universalis, « CELLULE - La division », sur Encyclopædia Universalis (consulté le )
- Jean-Yves Bouet, « Un nouvel éclairage sur la ségrégation de l’ADN chez les bactéries | INSB », sur www.insb.cnrs.fr (consulté le )
- (en) E. D. Goley, A. A. Iniesta et L. Shapiro, « Cell cycle regulation in Caulobacter: location, location, location », Journal of Cell Science, vol. 120, no 20, , p. 3501–3507 (ISSN 0021-9533 et 1477-9137, DOI 10.1242/jcs.005967, lire en ligne, consulté le )
- (en) Joe Lutkenhaus, Sebastien Pichoff et Shishen Du, « Bacterial cytokinesis: From Z ring to divisome », Cytoskeleton, vol. 69, no 10, , p. 778–790 (PMID 22888013, PMCID PMC3931253, DOI 10.1002/cm.21054, lire en ligne, consulté le )
- « La division asymétrique de certaines bactéries enfin comprise - Faculté de médecine - UNIGE », sur www.unige.ch, (consulté le )
- (en) Christos G Tsokos et Michael T Laub, « Polarity and cell fate asymmetry in Caulobacter crescentus », Current Opinion in Microbiology, vol. 15, no 6, , p. 744–750 (PMID 23146566, PMCID PMC3587792, DOI 10.1016/j.mib.2012.10.011, lire en ligne, consulté le )
- (en) Matthieu Bergé, Sébastien Campagne, Johann Mignolet et Seamus Holden, « Modularity and determinants of a (bi-)polarization control system from free-living and obligate intracellular bacteria », eLife, vol. 5, , e20640 (ISSN 2050-084X, PMID 28008852, PMCID PMC5182065, DOI 10.7554/eLife.20640, lire en ligne, consulté le )
- (en) Johann Mignolet, Seamus Holden, Matthieu Bergé et Gaël Panis, « Functional dichotomy and distinct nanoscale assemblies of a cell cycle-controlled bipolar zinc-finger regulator », eLife, vol. 5, , e18647 (ISSN 2050-084X, PMID 28008851, PMCID PMC5182063, DOI 10.7554/eLife.18647, lire en ligne, consulté le )
Liens externes modifier
- (en) Référence Catalogue of Life : Caulobacter vibrioides Henrici & Johnson, 1935 (consulté le )
- (fr + en) Référence EOL : Caulobacter vibrioides (consulté le )
- (fr + en) Référence GBIF : Caulobacter vibrioides A (consulté le )
- (en) Référence IRMNG : Caulobacter vibrioides Henrici & Johnson, 1935 (consulté le )
- (fr + en) Référence ITIS : Caulobacter vibrioides Henrici and Johnson, 1935 (consulté le )
- (en) Référence NCBI : Caulobacter vibrioides Henrici and Johnson 1935 (taxons inclus) (consulté le )
- (en) Référence Taxonomicon : Caulobacter vibrioides Henrici and Johnson 1935 (Approved Lists 1980) (consulté le )
- (en) Séquence du génome