Système de clivage de la glycine
Le système de clivage de la glycine (GCS), également appelé complexe de la glycine décarboxylase, est un système enzymatique intervenant en réponse à l'élévation de la concentration en glycine, un acide α-aminé protéinogène[1]. Il est parfois appelé glycine synthase lorsqu'il fonctionne dans le sens opposé pour produire de la glycine[2]. Présent à la fois chez les animaux, les plantes et les bactéries, il catalyse la réaction :
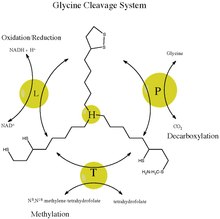
- glycine + tétrahydrofolate + NAD+ 5,10-méthylènetétrahydrofolate + CO2 + NH3 + NADH + H+.
Chez l'humain et la plupart des vertébrés, le système de clivage de la glycine constitue la principale voie métabolique de dégradation de la glycine et de la sérine. Ceci provient essentiellement du fait que cette réaction produit du 5,10-méthylènetétrahydrofolate, qui constitue l'un des rares donneurs de groupes monocarbonés pour les biosynthèses[2]. Le groupe méthyle issu de la glycine peut ainsi être transféré à d'autres molécules clés telles que des purines ou la méthionine.
Cette réaction intervient également dans la photorespiration chez les plantes. Le système de clivage de la glycine est présent en permanence dans les feuilles mais y demeure en faible quantités tant qu'elles ne sont pas exposées à la lumière. Lorsque la photosynthèse est à son maximum, les quantités de GCS augmentent d'un facteur 10[3].
Chez la bactérie anaérobie Clostridium acidiurici, le GCS fonctionne essentiellement dans le sens de la production de glycine. Bien que ce mode de fonctionnement soit également possible chez les animaux, il n'y est pas observé naturellement[4],[5].
Le système de clivage de la glycine est constitué de quatre protéines — la protéine P, la protéine T, la protéine L et la protéine H, — qui ne forment pas véritablement de complexe stable[6], de sorte que la désignation de « système » est plus appropriée que celle de « complexe ». La protéine H interagit avec les trois autres, qui sont des enzymes, comme support des produits intermédiaires de la décarboxylation de la glycine[2]. Ce système est lié à la membrane mitochondriale interne, aussi bien chez les animaux que chez les plantes. Des mutations affectant ce système sont liées à l'encéphalopathie glycinique[2].
| Enzyme ou protéine et abréviation(s) |
Numéro EC | Description |
| Protéine P (GLDC) |
EC | Glycine décarboxylase |
| Protéine T (GCST ou AMT) |
EC | Aminométhyltransférase |
| Protéine L (GCSL ou DLD) |
EC | Dihydrolipoyl déshydrogénase |
| Protéine H (GCSH) |
Protéine H du système de clivage de la glycine : possède un résidu d'acide lipoïque et interagit avec les trois enzymes du système de façon cyclique pour catalyser successivement une méthylamination réductrice par la glycine décarboxylase, un transfert de méthylamine par l'aminométhyltransférase et un transfert d'électron par la dihydrolipoyl déshydrogénase. | |
Le système de clivage de la glycine présente la composition P4T9L2H27.
Couplé à la sérine hydroxyméthyltransférase, il catalyse la réaction :
Le mécanisme réactionnel correspondant peut être représenté par le schéma ci-dessous :

La glycine, représentée en rouge, se condense avec le GCS (A) pour former une base de Schiff (B) qui réagit avec l'acide lipoïque de la protéine H après décarboxylation (C) et libération de dioxyde de carbone CO2.
Le groupe méthyle est transféré au tétrahydrofolate (1) pour former du 5,10-méthylènetétrahydrofolate (2) en libérant un ion ammonium NH4+.
L'acide dihydrolipoïque laissé par cette réaction (D) est oxydé par une molécule de FAD liée à la dihydrolipoyl déshydrogénase, molécule réduite en FADH2 (E) au cours de cette réaction puis régénérée par réduction d'une molécule de NAD+ pour redonner le GCS (A).
Le tétrahydrofolate est lui-même régénéré à partir du 5,10-tétrahydrofolate et d'une seconde molécule de glycine par la sérine hydroxyméthyltransférase avec formation de L-sérine.
Notes et références modifier
- (en) Goro Kikuchi, « The glycine cleavage system: Composition, reaction mechanism, and physiological significance », Molecular and Cellular Biochemistry, vol. 1, no 2, , p. 169-187 (lire en ligne) DOI 10.1007/BF01659328 ;
- (en) Goro KIKUCHI, Yutaro MOTOKAWA, Tadashi YOSHIDA et Koichi HIRAGA, « Glycine cleavage system: reaction mechanism, physiological significance, and hyperglycinemia », Proceedings of the Japan Academy, Series B, vol. 84, no 7, , p. 246-263 (PMCID 3666648, lire en ligne) DOI 10.2183/pjab.84.246 ;
- (en) David J. Oliver, Michel Neuburger, Jacques Bourguignon et Roland Douce, « Interaction between the Component Enzymes of the Glycine Decarboxylase Multienzyme Complex », Plant Physiology, vol. 94, no 2, , p. 833-839 (PMCID 1077305, lire en ligne) DOI 10.1104/pp.94.2.833 ;
- (en) R. T. Gariboldi et H. L. Drake, « Glycine synthase of the purinolytic bacterium, Clostridium acidiurici. Purification of the glycine-CO2 exchange system », Journal of Biological Chemistry, vol. 259, , p. 6085-6089 (lire en ligne)
- (en) G. Kikuchi et K. Hiraga, « The mitochondrial glycine cleavage system », Molecular and Cellular Biochemistry, vol. 45, no 3, , p. 137-149 (lire en ligne) DOI 10.1007/BF00230082 ;
- (en) Roland Douce, Jacques Bourguignon, Michel Neuburger et Fabrice Rébeillé, « The glycine decarboxylase system: a fascinating complex », Trends in Plants Sciences, vol. 6, no 4, , p. 167-176 (lire en ligne) DOI 10.1016/S1360-1385(01)01892-1 ;
