Malate déshydrogénase
| Malate déshydrogénase soluble | ||
| Caractéristiques générales | ||
|---|---|---|
| Nom approuvé | Malate déshydrogénase 1 | |
| Symbole | MDH1 | |
| N° EC | 1.1.1.37 | |
| Homo sapiens | ||
| Locus | 2p15 | |
| Masse moléculaire | 36 426 Da[1] | |
| Nombre de résidus | 334 acides aminés[1] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
| Malate déshydrogénase mitochondriale | ||
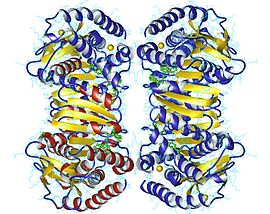 Tétramère de malate déshydrogénase mitochondriale humaine avec NAD en vert (PDB 2DFD). | ||
| Caractéristiques générales | ||
| Nom approuvé | Malate déshydrogénase 2 | |
| Symbole | MDH2 | |
| N° EC | 1.1.1.37 | |
| Homo sapiens | ||
| Locus | 7q11.23 | |
| Masse moléculaire | 35 503 Da[1] | |
| Nombre de résidus | 338 acides aminés[1] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
La malate déshydrogénase (MDH) est une oxydoréductase du cycle de Krebs qui catalyse la réaction :
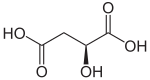
|
+ NAD+ NADH + H+ + | 
|
| L-Malate | Oxaloacétate |
Cette enzyme intervient dans plusieurs voies métaboliques majeures, notamment le cycle de Krebs.
| N° EC | EC |
|---|---|
| N° CAS |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
Isoformes modifier
Les procaryotes possèdent une seule forme, mais tous les eucaryotes en ont au moins deux isoformes, une dans le cytosol et l'autre dans la matrice mitochondriale[2]. Les mycètes et les plantes en ont également une forme spécifique aux glyoxysomes, qui intervient dans le cycle du glyoxylate. Les plantes en possèdent une quatrième forme spécifique aux chloroplastes et fonctionnant au NADP+, la malate déshydrogénase à NADP+ (EC ), qui intervient dans le cycle de Calvin, la fixation du carbone en C4 et le métabolisme acide crassulacéen.
Enfin, les archées possèdent une malate déshydrogénase étroitement apparentée à la lactate déshydrogénase.
Biochimie modifier
La malate déshydrogénase intervient en premier lieu dans le cycle de Krebs, où elle oxyde le L-malate en oxaloacétate. Ce dernier peut être à son tour condensé avec l'acétyl-CoA par la citrate synthase pour enclencher un nouveau tour du cycle de Krebs.
Elle intervient également dans la néoglucogenèse, sous ses formes mitochondriale et cytosolique. Le pyruvate peut en effet être converti en oxaloacétate par la pyruvate carboxylase, lequel est réduit en L-malate par la malate déshydrogénase mitochondriale fonctionnant alors en sens inverse de celui du cycle de Krebs : le L-malate peut quitter la mitochondrie pour rejoindre le cytosol et y être à nouveau oxydé en oxaloacétate par la malate déshydrogénase cytosolique. L'oxaloacétate est alors converti en phosphoénolpyruvate par la phosphoénolpyruvate carboxykinase (PEPCK).
La forme mitochondriale intervient également dans la navette malate-aspartate[3], essentielle à la respiration cellulaire.
Structure et mécanisme catalytique modifier
La malate déshydrogénase se présente, chez la plupart des êtres vivants, sous forme d'une protéine homodimérique dont la structure est étroitement apparentée à la lactate déshydrogénase. Il s'agit d'une grosse molécule dont les sous-unités ont une masse de 30 à 35 kDa. La séquence peptidique de ces enzymes tend à montrer qu'elles ont divergé en deux groupes phylogénétiques : les isoenzymes mitochondriales d'une part, et les isoenzymes cytosoliques et chloroplastiques d'autre part[4]. Ces observations corroborent la théorie endosymbiotique selon laquelle les mitochondries et les chloroplastes se sont développés à l'intérieur des cellules eucaryotes à partir de procaryotes ancestraux[5]. De plus, la séquence peptidique de la malate déshydrogénase des archées est plus proche de celle de la lactate déshydrogénase que de celle de la malate déshydrogénase des autres organismes, ce qui illustrerait une filiation entre la lactate déshydrogénase et la malate déshydrogénase[6].
Chaque sous-unité des dimères de malate déshydrogénase possède deux domaines distincts dont la structure et la fonctionnalité varie. Chaque domaine adopte une structure en feuillets β parallèles, avec quatre feuillets β et une hélice α comprenant le site de liaison central au NAD+. Les sous-unités sont liées entre elles par de nombreuses liaisons hydrogène et interactions hydrophobes[7].
Le site actif de la malate déshydrogénase est une cavité hydrophobe incluse dans la protéine dans laquelle se trouvent des sites de liaison spécifiques au substrat et à sa coenzyme, le NAD+. À l'état actif, l'enzyme subit une modification conformationnelle qui enferme le substrat afin de minimiser son exposition au solvant et de positionner à proximité du substrat les résidus d'acides aminés catalytiques clés[4]. Les trois résidus, qui comprennent une triade catalytique sont d'une part l'His195 et l'Asp168, qui agissent tous les deux comme système de transfert de proton, et les résidus Arg102, Arg109 et Arg171, qui fixent le substrat[8]. Les études cinétiques ont montré que l'activité enzymatique de la malate déshydrogénase est séquencée : le NAD+/NADH est lié avant le substrat[9].
Notes et références modifier
- Les valeurs de la masse et du nombre de résidus indiquées ici sont celles du précurseur protéique issu de la traduction du gène, avant modifications post-traductionnelles, et peuvent différer significativement des valeurs correspondantes pour la protéine fonctionnelle.
- (en) P. Minárik, N. Tomásková, M. Kollárová et M. Antalík, « Malate dehydrogenases – structure and function », General Physiology and Biophysics, vol. 21, no 3, , p. 257-265 (PMID 12537350, lire en ligne)
- (en) R. A. Musrati, M. Kollárová, N. Mernik et D. Mikulásová, « Malate dehydrogenase: distribution, function and properties », General Physiology and Biophysics, vol. 17, no 3, , p. 193-210 (PMID 9834842, lire en ligne)
- (en) Christopher R. Goward et David J. Nicholls, « Malate dehydrogenase: A model for structure, evolution, and catalysis », Protein Science, vol. 3, no 10, , p. 1883-1888 (PMID 7849603, PMCID 2142602, DOI 10.1002/pro.5560031027, lire en ligne)
- (en) Lee McAlister-Henn, « Evolutionary relationships among the malate dehydrogenases », Trends in Biochemical Sciences, vol. 13, no 5, , p. 178-181 (PMID 3076279, DOI 10.1016/0968-0004(88)90146-6, lire en ligne)
- (en) Fabrice Cendrin, Jadwiga Chroboczek, Giuseppe Zaccai, Henryk Eisenberg et Moshe Mevarech, « Cloning, sequencing, and expression in Escherichia coli of the gene coding for malate dehydrogenase of the extremely halophilic archaebacterium Haloarcula marismortui », Biochemistry, vol. 32, no 16, , p. 4308-4313 (PMID 8476859, DOI 10.1021/bi00067a020, lire en ligne)
- (en) Michael D. Hall, David G. Levitt et Leonard J. Banaszak, « Crystal structure of Escherichia coli malate dehydrogenase: A complex of the apoenzyme and citrate at 1·87 Å resolution », Journal of Molecular Biology, vol. 226, no 3, , p. 867-882 (PMID 1507230, DOI 10.1016/0022-2836(92)90637-Y, lire en ligne)
- (en) Victor S. Lamzin, Zbigniew Dauter et Keith S. Wilson, « Dehydrogenation through the looking-glass », Nature Structural Biology, vol. 1, no 5, , p. 281-282 (PMID 7664032, DOI 10.1038/nsb0594-281, lire en ligne)
- (en) Thomas B. Shows, Verne M. Chapman et Frank H. Ruddle, « Mitochondrial malate dehydrogenase and malic enzyme: Mendelian inherited electrophoretic variants in the mouse », Biochemical Genetics, vol. 4, no 6, , p. 707-718 (PMID 5496232, DOI 10.1007/BF00486384, lire en ligne)
