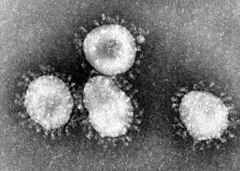
Coronavirus de la gastro-entérite transmissible porcine
Coronavirus
- Espèce : Alphacoronavirus 1
- Coronavirus de la gastro-entérite transmissible porcine (TGEV, porc)
- Coronavirus félin (chat)
- Coronavirus canin (chien)
Le Coronavirus de la gastro-entérite transmissible (TGEV, pour "Transmissible gastroenteritis coronavirus)" est un coronavirus qui infecte les porcs. Il s'agit d'un virus à ARN simple brin enveloppé, de sens positif, qui pénètre dans sa cellule hôte en se liant au récepteur APN[1]. Le virus appartient au genre Alphacoronavirus, sous-genre Tegacovirus, espèce Alphacoronavirus 1[2],[3].
Les protéines qui contribuent à la structure globale du TGEV comprennent la pointe (S), l'enveloppe (E), la membrane (M) et la nucléocapside (N). Le génome des coronavirus comprend environ 28,6 nucléotides. D'autres coronavirus appartenant à l'espèce Alphacoronavirus 1 sont le coronavirus félin, le coronavirus canin et le virus de la péritonite infectieuse féline .
Taxonomie modifier
Le TGEV appartient à la famille Coronaviridae, genre Alphacoronavirus, espèce Alphacoronavirus 1. Il s'agit d'un virus enveloppé avec un génome à ARN simple brin positif. Le TGEV possède trois protéines structurales principales: la phosphoprotéine (N), la protéine membranaire intégrale (E1) et la grande glycoprotéine (E2). La protéine N encapsule l'ARN génomique et la protéine S forme des projections virales.
Morphologie modifier
La morphologie du TGEV a été principalement déterminée par des techniques de microscopie électronique[4]. La morphologie est similaire à celle du myxovirus, en ce qu'ils ont des projections de surface et une enveloppe. Les virus sont principalement de forme circulaire avec un diamètre allant de 100 à 150 nm, y compris les projections de surface.
Pathologie modifier
Le TGEV infecte les porcs. Chez les porcelets de moins d'une semaine, le taux de mortalité est proche de 100%. La pathologie du TGEV est similaire à celle des autres coronavirus. Une fois que le virus infecte l'hôte, il se multiplie dans la paroi cellulaire de l'intestin grêle, entraînant la perte de cellules absorbantes, ce qui entraîne à son tour un raccourcissement des villosités. Les porcs infectés ont alors une capacité réduite à digérer les aliments et meurent de déshydratation[5].
Le TGEV était répandu aux États-Unis lorsqu'il a été découvert à l'origine au début du XXe siècle. Il est devenu plus rare à la fin des années 1980 avec la montée du coronavirus respiratoire porcin (PRCV). On pense que le PRCV confère une certaine immunité au TGEV[6].
Biologie[7] modifier
Les coronavirus pénètrent dans l'hôte en se fixant d'abord à la cellule hôte à l'aide de la glycoprotéine "Spike" qui forme des péplomères à la surface du virion. La protéine Spike interagit avec l'aminopeptidase porcine N (pAPN), qui joue le rôle de récepteur cellulaire, pour faciliter l'entrée du virus dans la cellule. Le même récepteur cellulaire est également un point de contact pour certains coronavirus humains.
La protéine E2 forme une projection d'environ 20 nm sur la surface du virion, en forme de pétale. On pense que la protéine E2 est impliquée dans la pathogenèse en aidant le virus à pénétrer dans le cytoplasme de l'hôte. La protéine E2 a initialement 1447 acides aminés, avant clivage d'une courte séquence hydrophobe. Après glycosylation de la protéine dans l'appareil de Golgi, la protéine est ensuite incorporée au nouveau virion. Il existe plusieurs domaines fonctionnels au sein de la protéine E2: un segment hydrophobe de 20 résidus à l'extrémité C-terminale ancre la protéine dans la membrane lipidique; le reste de la protéine est divisé en deux parties: un tronçon hydrophile qui se trouve à l'intérieur du virion et un tronçon riche en Cystéine qui sont probablement des sites d'acylation.
La protéine E1 est principalement intégrée dans l'enveloppe lipidique du virion et joue donc un rôle essentiel dans l'architecture du virus. On pense que la protéine E1 interagit avec la membrane lymphocytaire, ce qui conduit à l'induction de gènes codant l'IFN.
Le TGEV en biotechnologies modifier
Le TGEV a été utilisé comme vecteur d'expression[8]. Le vecteur a été construit en remplaçant les cadres ouverts de lecture non essentiels 3a et 3b, sous le contrôle de séquences de régulation de la transcription (TRS) par la protéine fluorescente verte (GFP). La construction résultante était encore entéropathogène, mais avec une multiplication réduite. L'infection des cellules par ce virus altéré provoque une réponse immunitaire spécifique contre la protéine hétérologue. Ce vecteur est utilisé dans le développement d'un vaccin voire d'une thérapie génique. La motivation pour l'ingénierie du génome du TGEV est que les coronavirus ont de grands génomes, donc ils ont de la place pour l'insertion de gènes étrangers. Les coronavirus infectent également les voies respiratoires et peuvent être utilisés pour cibler des antigènes sur cette zone et générer une réponse immunitaire.
Voir également modifier
Liens internes modifier
Références modifier
- (en) Anthony R. Fehr et Stanley Perlman, « Coronaviruses: An Overview of Their Replication and Pathogenesis », dans Coronaviruses, vol. 1282, Springer New York, (ISBN 978-1-4939-2437-0, PMID 25720466, PMCID PMC4369385, DOI 10.1007/978-1-4939-2438-7_1, lire en ligne), p. 1–23
- (en) Patrick C. Y. Woo, Yi Huang, Susanna K. P. Lau et Kwok-Yung Yuen, « Coronavirus Genomics and Bioinformatics Analysis », Viruses, vol. 2, no 8, , p. 1804–1820 (ISSN 1999-4915, PMID 21994708, PMCID PMC3185738, DOI 10.3390/v2081803, lire en ligne, consulté le )
- « Taxonomy browser (Alphacoronavirus 1) », www.ncbi.nlm.nih.gov (consulté le )
- (en) M. Tajima, « Morphology of transmissible gastroenteritis virus of pigs: A possible member of coronaviruses », Archiv für die gesamte Virusforschung, vol. 29, no 1, , p. 105–108 (ISSN 0304-8608 et 1432-8798, PMID 4195092, PMCID PMC7086923, DOI 10.1007/BF01253886, lire en ligne, consulté le )
- Harris, « Transmissible Gastroenteritis in Pigs », Merck Veterinary Manual, Merck (consulté le )
- Transboundary and Emerging Diseases of Animals, Iowa State University, (ISBN 978-0-9846270-5-9)
- (en) Hubert Laude, Denis Rasschaert, Bernard Delmas et Murielle Godet, « Molecular biology of transmissible gastroenteritis virus », Veterinary Microbiology, vol. 23, nos 1-4, , p. 147–154 (PMID 2169670, PMCID PMC7117338, DOI 10.1016/0378-1135(90)90144-K, lire en ligne, consulté le )
- (en) Isabel Sola, Sara Alonso, Sonia Zúñiga et Mónica Balasch, « Engineering the Transmissible Gastroenteritis Virus Genome as an Expression Vector Inducing Lactogenic Immunity », Journal of Virology, vol. 77, no 7, , p. 4357–4369 (ISSN 0022-538X et 1098-5514, PMID 12634392, PMCID PMC150661, DOI 10.1128/JVI.77.7.4357-4369.2003, lire en ligne, consulté le )